Introduction to data visualization [FR]
R-plot & ggplot2
Skills
- Teaching
- R
- Data viz
TP Visualisation avec R
Avant le cours
Dans une publication scientifique (un article, un livre) repérez une figure que vous trouvez excellente (claire, pertinente, esthétique) et une figure que vous ne trouvez pas bien (pas claire, moche, illisible) et argumentez votre choix.
##Installation du RStudio Desktop
Allez sur le site http://www.rstudio.com/products/rstudio/download/ et téléchargez le logiciel.
Si l’installation n’est pas possible, allez dans la section sans R studio.

Premiers pas avec RStudio
File-> New File->Script
Enregistrer le nouveau script directement en cliquant sur la petite icone de disquette. N’oubliez pas de sauvegarder votre script régulièrement (Ctr+S).
Vous devriez voir votre écran divise en 4 parties principales: Script, Console, Environment/History et Files/Plots/Packages/Help/Viewer.
Dans la partie script vous écrivez les commandes et pour les faire executer vous allez dans Code->Run.
Ou bien sélectionnez lines que vous voulez executer et tapez Ctr+Enter.
Essayez
print("Hello world") #[Ctr+Enter][1] “Hello world”
Si vous cliquez sur History vous devriez voir:
print('Hello world')
Historique sauvegarde les commandes effectuées dans l’ordre hronologique.
Pour savoir plus sur la fonction print
tapez:
?printdirectement dans la console, a votre droite l’onglet Help va afficher directement les informations sur print.
Dans le R, toutes les fonctions sont bien documentées. A la fin des informations sur la fonction vous allez toujours trouver les exemples d’utilisation.
Sans R studio - the brute
Vous allez executer les scripts R directement dans la ligne de commandes de votre Unix/Mac.
Ouvrez terminal et tapez: R
Cela va ouvrir le logiciel directement dans le terminal.
Vous pouvez taper print('Hello world') directemment dans la console.
Vous allez voir
> print('Hello world')
[1] "Hello world"
Pour l’aide sur la fonction tapez:
?print
(marche avec toutes les fonctions)
Dans R, toutes les fonctions sont bien documentées. A la fin des informations sur la fonction vous allez toujours trouver les exemples d’utilisation.
Neanmoins, pour garder tous vos commandes dans un document, et effectuer les commandez plus complexes, sauvgarder votre commande dans un fichier et effectuez avec R.
- tapez gedit (Unix) ou un autre editeur de texte simple (pas MS Word)
- écrivez
print('Hello world') - sauvgarder le fichier myscript.R
- dans le terminal, dans le même répertoire où vous avez sauvegardé le fichier tapez
Rscript myscript.R
Dans la console vous devriez voir
[1] "Hello world"

##Manipulation de données
Importez les données
Enregistrez le fichier ozone.txt http://www.agrocampus-ouest.fr/math/livreR/ozone.txt sur votre ordinateur.
Ensuite importez dans R, pour se faciliter la tâche decidez dans quel repertoire voulez avoir vos fichiers R de cette session
setwd('/chemin')par example si le fichier est dans ~/Downloads:
setwd('/XXXX/xxxx/Downloads')pour connaitre le chemin complet de votre fichier ozone.txt, allez dans le repértoire où il est enregistré et tapez dans le terminal:
pwd
Ensuite dans le script (fenêtre Script dans Rstudio ou votre fichier .R):
read.table("ozone.txt", sep=" ", header=TRUE)Quoique, vous devriez éviter d’afficher les données dans la console. Dans le cas de grands jeux de données cela peut ralentir énormément le système. Donc mieux c’est:
mytable=read.table("ozone.txt", sep=" ", header=TRUE)Dès que vous créez une variable, elle apparait dans l’onglet environnement et vous pouvez voir ses caractéristiques (RStudio).
La bonne pratique: Après avoir importé votre jeu de données, vérifiez toujours s’il est bien importé. Une manière simple, c’est de faire:
summary(mytable)## maxO3 T9 T12 T15
## Min. : 42.00 Min. :11.30 Min. :14.00 Min. :14.90
## 1st Qu.: 70.75 1st Qu.:16.20 1st Qu.:18.60 1st Qu.:19.27
## Median : 81.50 Median :17.80 Median :20.55 Median :22.05
## Mean : 90.30 Mean :18.36 Mean :21.53 Mean :22.63
## 3rd Qu.:106.00 3rd Qu.:19.93 3rd Qu.:23.55 3rd Qu.:25.40
## Max. :166.00 Max. :27.00 Max. :33.50 Max. :35.50
## Ne9 Ne12 Ne15 Vx9
## Min. :0.000 Min. :0.000 Min. :0.00 Min. :-7.8785
## 1st Qu.:3.000 1st Qu.:4.000 1st Qu.:3.00 1st Qu.:-3.2765
## Median :6.000 Median :5.000 Median :5.00 Median :-0.8660
## Mean :4.929 Mean :5.018 Mean :4.83 Mean :-1.2143
## 3rd Qu.:7.000 3rd Qu.:7.000 3rd Qu.:7.00 3rd Qu.: 0.6946
## Max. :8.000 Max. :8.000 Max. :8.00 Max. : 5.1962
## Vx12 Vx15 maxO3v vent pluie
## Min. :-7.878 Min. :-9.000 Min. : 42.00 Est :10 Pluie:43
## 1st Qu.:-3.565 1st Qu.:-3.939 1st Qu.: 71.00 Nord :31 Sec :69
## Median :-1.879 Median :-1.550 Median : 82.50 Ouest:50
## Mean :-1.611 Mean :-1.691 Mean : 90.57 Sud :21
## 3rd Qu.: 0.000 3rd Qu.: 0.000 3rd Qu.:106.00
## Max. : 6.578 Max. : 5.000 Max. :166.00Pour connaître les dimensions du tableau
dim(mytable)## [1] 112 13A quoi corresponds premier chiffre? deuxième chiffre?

Organisation d’information:
head(mytable)## maxO3 T9 T12 T15 Ne9 Ne12 Ne15 Vx9 Vx12 Vx15 maxO3v
## 20010601 87 15.6 18.5 18.4 4 4 8 0.6946 -1.7101 -0.6946 84
## 20010602 82 17.0 18.4 17.7 5 5 7 -4.3301 -4.0000 -3.0000 87
## 20010603 92 15.3 17.6 19.5 2 5 4 2.9544 1.8794 0.5209 82
## 20010604 114 16.2 19.7 22.5 1 1 0 0.9848 0.3473 -0.1736 92
## 20010605 94 17.4 20.5 20.4 8 8 7 -0.5000 -2.9544 -4.3301 114
## 20010606 80 17.7 19.8 18.3 6 6 7 -5.6382 -5.0000 -6.0000 94
## vent pluie
## 20010601 Nord Sec
## 20010602 Nord Sec
## 20010603 Est Sec
## 20010604 Nord Sec
## 20010605 Ouest Sec
## 20010606 Ouest PluieCela vous donnée une idée comment sont les premières 6 lignes du tableau.
1.Afficher juste la première colonne du tableau ‘mytable’
2.Afficher juste la première ligne du tableau ‘mytable’
3.Afficher les noms de colonnes du tableau ‘mytable’
4.Créez un vecteur ‘Group’ de la meme longueur qu’une colonne du tableau contenant le chiffre 1 pour la moitié de lignes et le chiffre 2 l’autre moitié (indice function ‘rep’)
5.l’ajoutez au tableau comme une colonne (indice function ‘cbind’)
6.Supprimez la colonne que vous venez d’ajouter
Pour répondre aux questions utilisez help("fonction") ou ?fonction, Google etc.

Visualisation des données R standard
Pour RStudio
Commencer par la commande
demo(graphics)et observez l’onglet Plots Dans la console s’affichent les commandes utilisées pour obtenir les graphs.
Sans R studio
- Ouvrir R session dans le terminal (
R) - Tapez directement
demo(graphics)- Tapez la touche
Entrée - Une fênetre blanche va s’ouvrir, revenez dans le terminal et tapez la touche
Entrée***
Mais commençant par nos donnés:
Le jeu de donnés contient les variables climatiques et une variable de pollution à l’ozone mesurées durant l’été 2001 à Rennes. Les variables considérées ici seront:
max03 - maximum de l’ozone journalier
T12 - température à midi
vent - direction du vent
pluie
Vx12 - projection du vecteur vitesse du vent sur l’axe Est-Ouest
Regardez encore une fois
summary(mytable) Lesquelles variables sont quantitatives? Qualitatives?
Sélectionnez seulement les colonnes mentionnez et enregistrez dans une variable ozon
ozone=mytable[,c('T12','maxO3','vent','pluie','Vx12')]
summary(ozone)## T12 maxO3 vent pluie Vx12
## Min. :14.00 Min. : 42.00 Est :10 Pluie:43 Min. :-7.878
## 1st Qu.:18.60 1st Qu.: 70.75 Nord :31 Sec :69 1st Qu.:-3.565
## Median :20.55 Median : 81.50 Ouest:50 Median :-1.879
## Mean :21.53 Mean : 90.30 Sud :21 Mean :-1.611
## 3rd Qu.:23.55 3rd Qu.:106.00 3rd Qu.: 0.000
## Max. :33.50 Max. :166.00 Max. : 6.578Afin de représenter deux colonnes comme un nuage de points. Observez le taux d’O3 maximale en fonction de la température à midi
#plot(x,y)
plot(ozone[,'T12'],ozone[,'maxO3'])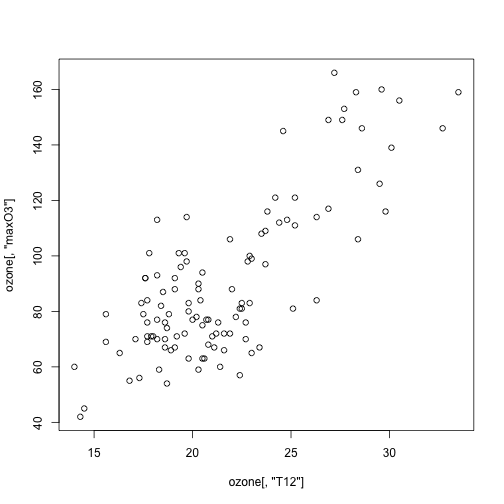
ou bien
plot(maxO3~T12, data=ozone)Visualisez le taux maximal d’O3 (maxO3) en fonction de la variable vent poursuivant la même logique
plot(maxO3~vent, data=ozone,xlab='Secteur du vent', ylab="pic d'ozone")ici equivalent à
boxplot(maxO3~vent, data=ozone,xlab='Secteur du vent', ylab="pic d'ozone")Annotez les parties de la boite à moustaches (boxplot).
Vous pouvez aussi personnaliser le graph.
Ajoutez les couleurs:
col = c("blue", "red", "green", "yellow")
boxplot(maxO3 ~ vent, data = ozone, xlab = "Secteur du vent", ylab = "pic d'ozone",
col = c("blue", "red", "green", "yellow"))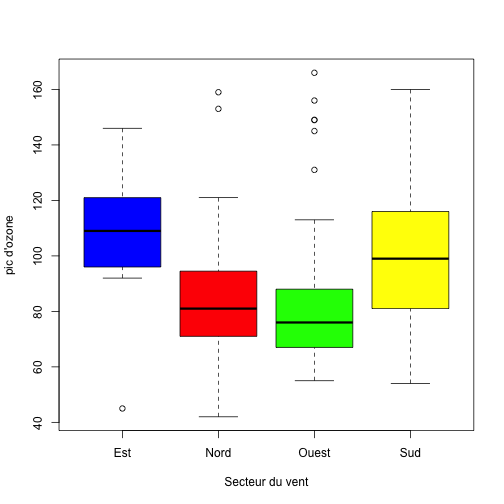
Si vous avez de difficultés à comprendre ce qui répresentent ‘boxplot’ faites une exercice:
Si l’on s’intéresse qu’à la catégorie l’Est
Selectionnez une partie du tableau qui correspond au vent de l’Est
ozoneE=ozone[ozone$vent=='Est',]Faites une boite à moustache
boxplot(ozoneE[,2],xlab='Secteur du vent', ylab="pic d'ozone") 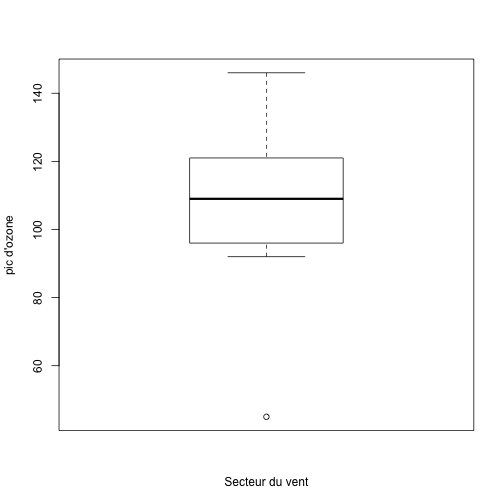
observez les valeurs dans l’ordre
sort(ozoneE$maxO3)## [1] 45 92 96 98 106 112 114 121 126 146Calculez la moyenne
mean(ozoneE$maxO3)## [1] 105.6Calculez les quantiles/ a quoi correspond-ils?
quantile(ozoneE$maxO3)## 0% 25% 50% 75% 100%
## 45.00 96.50 109.00 119.25 146.00Essayez maintenant d’observer ces éléments sur le graph
Pour visualiser deux variables qualitatives essayez:
plot(pluie~vent, data=ozone)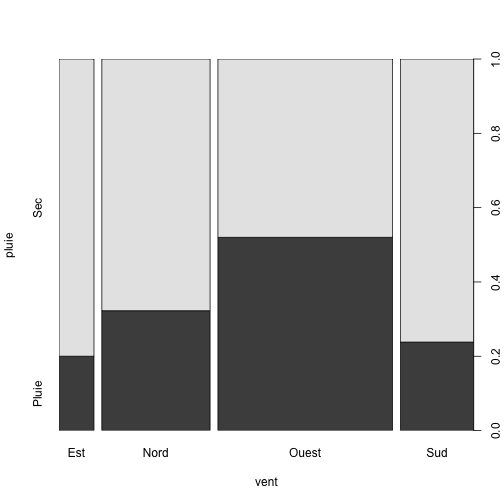
Pareil, faites un graphe de direction du vent en fonction de température
Distribution
hist(ozone$maxO3,xlab='Ozone',main='Histogram')Décrivez le graph, en quels unités est l’axe y?
Utilisez les informations trouvé sur le site http://www.statmethods.net/advgraphs/parameters.html pour transformer en jouant avec les paramètres.
plot(maxO3~T12, data=ozone)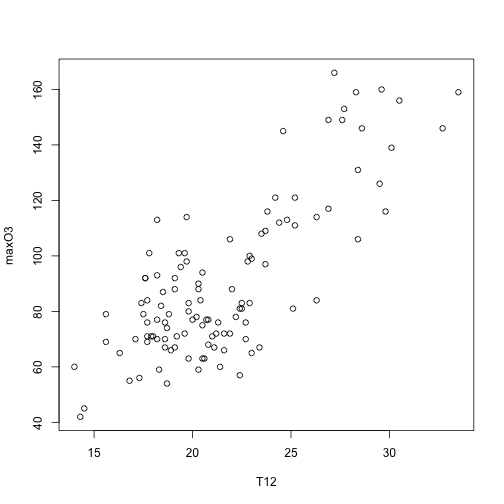
en
Notez le code R dans le compte-rendu des TD
GGPLOT2 - ajoutons une couche
Pour installer effectuez la commande:
install.packages("ggplot2") ## Installing package into '/Users/urszulaczerwinska/Library/R/3.3/library'
## (as 'lib' is unspecified)##
## The downloaded binary packages are in
## /var/folders/mz/7mx96rvs3xdchysdyy5w37b80000gq/T//RtmpGPMlST/downloaded_packageset ensuite
library(ggplot2)Dans le concept de ggplot2 le graphs sont compose de différents couches superposées.
En utilisant le même jeu de données tapez
ggplot(ozone, aes(x = T12, y = maxO3))R vous informe que le graphe n’a pas de couches
sauvegardez le plot dans un objet p et ajoutez une couche
Scatterplot</h3>
p <- ggplot(ozone, aes(x = T12, y = maxO3))
p <- p + geom_point()
p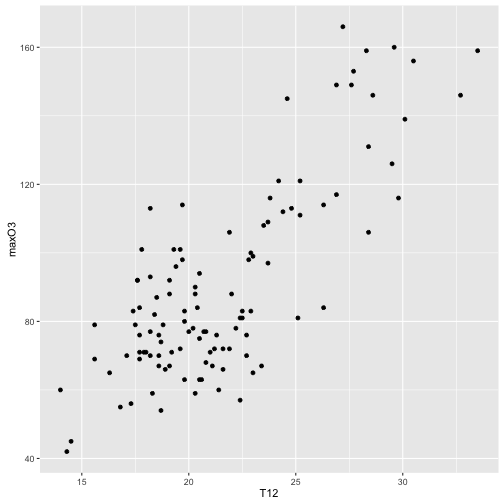
Faites attention au operateur + qui va servir toujours a ajouter les couches. Chaque nouvelle couche est une fonction qui prend en argument ses caractéristiques
Essayez
ggplot(ozone, aes(x = T12, y = maxO3)) + geom_point(color = "red", size = 5)Une couche statistique
p <- p + stat_smooth()La ligne représente un ‘fit’ et la bande grise, elle répresente l’intervalle de confiance établie en utilisant la méthode loess
Vous pouvez aussi visualiser la ligne sans points
ggplot(ozone, aes(x = T12, y = maxO3)) + stat_smooth()Pour bien annoter le graph:
p <- ggplot(ozone, aes(x = T12, y = maxO3)) + geom_point(color = "red", size = 5) +
ylab("taux maximal d'O3") + xlab("temperature a midi") + theme_bw() + labs(title = "taux d'ozone en fonction de la temperature a Rennes")Que fait-il theme_bw()?
C’est facile aussi de colorier les points en fonction du type de vent
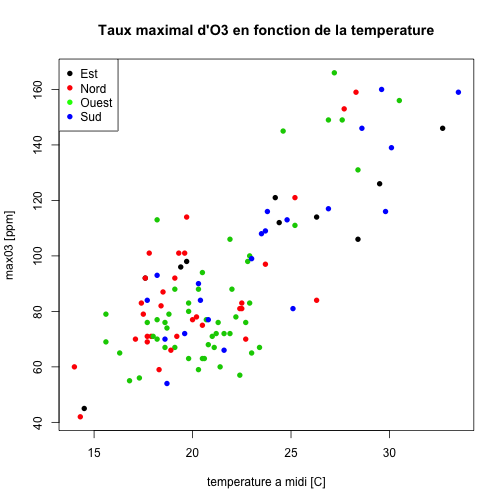
p <- ggplot(ozone, aes(x = T12, y = maxO3, color = vent)) + geom_point(size = 3) +
ylab("taux maximal d'O3") + xlab("temperature a midi") + theme_bw() + labs(title = "taux d'ozone en fonction de la temperature a Rennes")Observez que la légende apparait toute seule!
Vous pouvez joindre les points avec les lignes par groupe aussi
p <- ggplot(ozone, aes(x = T12, y = maxO3, color = vent)) + geom_point(size = 3) +
ylab("taux maximal d'O3") + xlab("temperature a midi") + theme_bw() + labs(title = "taux d'ozone en fonction de la temperature a Rennes") +
geom_line()Il est aussi possible de mapper les couleurs de valeurs continues
p <- ggplot(ozone, aes(x = T12, y = maxO3, color = maxO3)) + geom_point(size = 3) +
ylab("taux maximal d'O3") + xlab("temperature a midi") + theme_bw() + labs(title = "taux d'ozone en fonction de la temperature a Rennes")BARPLOT
p <- ggplot(ozone, aes(x =vent))+geom_bar()avec les couleurs
p <- ggplot(ozone, aes(x =vent, fill=vent))+geom_bar()Pour changer la palette de coloration ajoutez
p+
scale_fill_brewer(palette = "Set1")Ou décidez quels couleurs vous preferez pqr vous memes
p+
scale_fill_manual(values=c("bisque", "chartreuse4",
"hotpink","yellow"))error bars
ggplot(ozone, aes(vent, maxO3,fill=vent))+
stat_summary(fun.y = mean, geom = "bar")+
stat_summary(fun.data = mean_sdl, geom = "errorbar")## Warning: Computation failed in `stat_summary()`:
## Hmisc package required for this functionmean_sdl- retourne la moyenne du groupe, et les error bars qui correspond a l’ecart-type
vous pouvez essayer aussi
mean_cl_boot()
mean_cl_normal()
median_hilow()
Documentation:
● mean_cl_boot()
This will return the sample mean, and 95% bootstrap confidence intervals.
● mean_cl_normal()
This will return the sample mean, and the 95% percent Gaussian confidence interval based on the t-distribution
● mean_sdl()
This will return the sample mean and values at 1 sd and -1 sd away. You can make it return points any arbitrary number of sds away by passing that value to mult. For example, mult = 2 will return 2 and -2 sds.
● median_hilow()
This will return the sample median, and confidence intervals running from the 0.025 quantile to the 0.975 quantile, which covers 95% of the range of the data. You can change what range of the data you want the confidence interval to cover by passing it to conf.int. For example conf.int = 0.5 will return confidence intervals ranging from the 0.25 quantile to the 0.75 quantile.
Densité
Executez
ggplot(ozone, aes(maxO3, T12))+
stat_density2d()+geom_point()Vous pouvez jouer avec la coloration
ggplot(ozone, aes(maxO3, T12))+
stat_density2d(geom = "point",contour = F,
aes(size = ..density..), alpha = 0.3)ggplot(ozone, aes(maxO3, T12))+
stat_density2d(geom = "tile", contour = F, aes(fill = ..density..))ou bien
ggplot(ozone, aes(maxO3, T12)) + stat_density2d(geom = "tile", contour = F,
aes(fill = ..density..)) + scale_fill_gradientn(colours = rainbow(6))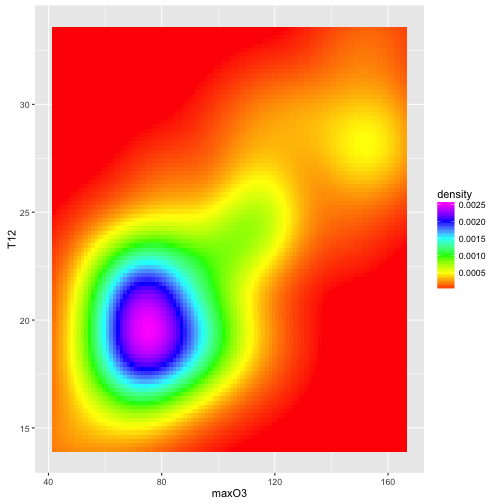
Ressources:
handout_ggplot2.pdf
http://www.ling.upenn.edu/~joseff/avml2012/#Section_1
http://www.cookbook-r.com/Graphs/Bar_and_line_graphs_%28ggplot2%29/
Les réponses diponibles à la demande
Le travail effectué pour Licence Frontières du Vivant at Center for Interdisciplinary Reserch.